|
|
BMe Kutatói pályázat |
|
Oláh György Doktori Iskola
BME VBK / Alkalmazott Biotechnológia és Élelmiszertudományi Tanszék
Témavezető: Dr. Vértessy G. Beáta
A DNS-beli uracil kvantifikálása egy érzékeny jelölő módszerrel
A kutatási téma néhány soros bemutatása
A genom metabolizmus manapság egy nagyon kutatott téma, mi sem jelzi ezt jobban, minthogy 2015-ben a Kémiai Nobel-díjat DNS-javításhoz kapcsolódó kutatásokért adták. A sejtek kromoszómaállománya nap mint nap olyan endogén és exogén támadásoknak van kitéve, mint az UV-sugárzás, a szabad gyökök és a karcinogén ágensek. A DNS sérülhet sejtosztódáskor, és a sejt saját metabolizmusa során. Így a DNS-hibajavításnak kiemelt jelentősége van minden élő szervezetben. Az uracil az egyik leggyakoribb DNS-hiba. A hidrolitikus citozin dezamináció, amely uracilt eredményez, 100–500 alkalommal történik meg egy emlős genomban naponta [1], és ha nincs kijavítva, pontmutáció jön létre. A pontmutációk rákot idézhetnek elő, és szerepük van az öregedésben is.
A kutatóhely rövid bemutatása
Vértessy G. Beáta kutatócsoportja részben a BME-n, részben, az MTA TTK-ban van: Biostruct Laboratory (BME) és Genom Metabolizmus Kutatócsoport (MTA TTK). A Vértessy kutatócsoport főbb kutatási területei a fehérje krisztallográfia és szerkezetmegfejtés; valamint fehérje-fehérje és fehérje-DNS kölcsönhatás vizsgálat in vitro és in vivo emlős sejtekben és baktériumokban.
A kutatás történetének, tágabb kontextusának bemutatása
A dezoxiuridin az egyik leggyakoribb DNS hiba. Az uracil az egyike az RNS-t felépítő négy heterociklusos bázisnak, azonban a DNS-ben a kémiailag kevésbé stabil uracil helyett timin a negyedik építőkő (1. ábra). Az uracil a DNS-ben két módon jelenhet meg: a DNS polimerázok beépíthetik timin helyett, vagy a citozin dezamináció eredményeképpen. A DNS-hibajavítás [S1] egyik útvonala a báziskivágó javítás (BER). A BER és a nukleotidszintek szabályozása felelősek a DNS-beli uracil felhalmozódás megakadályozásáért. Ebben a folyamatban a két kulcsenzim az uracil-DNS glikoziláz (UDG) és a dUTPáz [S2,S3]. Az uracil azonban fiziológiás körülmények között is megjelenhet a DNS-ben, úgy mint a magas uraciltartalmú bakteriofágok [2,3], és a HIV vírus genom esetében [4].
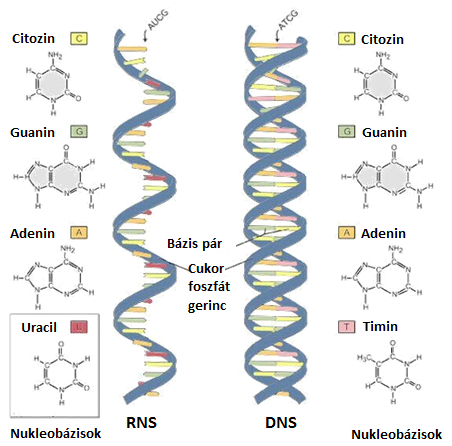
1. ábra: Nukleobázisok az RNS-ben és a DNS-ben
A DNS-beli uracil normál intermedier a szerzett immunitásban a humán B limfocitákban [5], valamint emelkedett a szintje az ecetmuslica lárvákban, bábokban és imagoban [6]. A rák kezelésben használt kemoterápiás szemlélet esetében is fontos a genomi uracil megjelenése. Az irodalomban több különböző specificitású, érzékenységű és árú genomi uracil kvantifikáló módszer ismert [7, 8, 9], amelyeket az 1. táblázatban foglaltam össze.
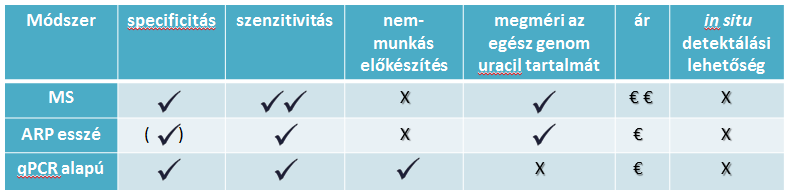
1. táblázat: Az uracil kvantifikáló módszerek összehasonlítása
Az MS-alapú módszer érzékeny, de igen drága műszert és magas szaktudást igényel. Az aldehid reaktív próba (ARP) esszé az UDG családba tartozó UNG enzim által létrehozott abázikus helyet méri. Egy kvantitatív qPCR alapú módszer relatív mennyiség meghatározásra alkalmas.
A kutatás célja, a megválaszolandó kérdések
Munkám során azt tűztem ki célul, hogy egy megbízható, gyors, olcsó és egyszerű módszert hozzak létre, amely kvantitatív és kvalitatív DNS-beli uracil meghatározásra is alkalmas. Egy olyan uracilszenzort fejlesztettünk ki egy katalitikusan inaktív mutáns UNG-ot felhasználva, amely képes kötni az uracilt úgy, hogy lehasítani már nem képes azt [S4]. A szenzorunk lehetővé tesz in situ immunocitokémiás felhasználást is. Elsőként azt akartam bizonyítani, hogy uracil szenzorunk valóban specifikusan felismeri az uracilt, de nem hasítja el az N-glikozidos kötést. Majd validálni kellett a módszert egy megfelelő standard és ismeretlen uraciltartalmú genomi DNS-minták felhasználásával.
Módszerek
Az új uracilkvantifikáló módszerhez egy katalitikusan inaktív UNG-ot használtam. Az uracilkötő UNG-szenzort úgy terveztük, hogy konvencionális antitestekkel lehessen detektálni dot blot alkalmazásokban és in situ immuncitokémiás kimutatások során is. A módszer sémája a 2. ábrán látható. Először a DNS-t nitrocellulóz membránra immobilizálom, és katalitikusan inaktív, Flag fúziós címkét tartalmazó katalitikusan inaktív UNG-gal jelölöm, majd elsődleges (anti-Flag) és másodlagos (tormaperoxidázzal kapcsolt anti-egér) antitestet kapcsolok a komplexhez. Megnövelt kemilumineszcencia jelet lehet detektálni.
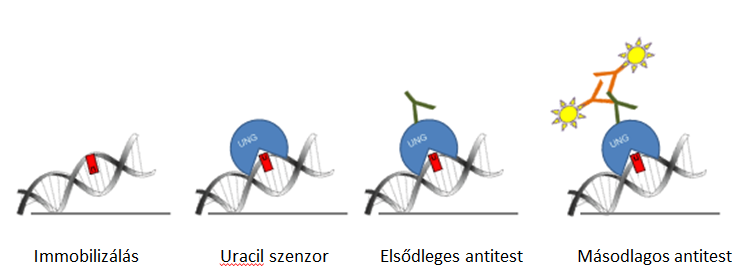
2. ábra A (dot blot-alapú) genomi uracil kvantifikáló módszer sémája
UNG-aktivitás-esszét alkalmaztunk, hogy megmutassuk, hogy a dupla mutáns (D154N, H277N) UNG-konstrukciók (a két pontmutáció hatására) nem rendelkeznek uracilkivágó aktivitással uracilgazdag DNS-en. Magas uraciltartalmú, illetve “normális” plazmid DNS-t kezeltem vad típusú (vt) vagy mutáns UNG-konstrukcióval, amit a bázismentes helyen a DNS-gerincet elhasító AP endonukleáz kezelés követett. A mintákat agaróz gélelektroforézissel analizáltam. A dot blot során használt 3xFLAG-ΔUNG- konstrukció uracilkötő képességét elektroforetikus mobilitáseltolódás-esszével vizsgáltam. Ha az UNG- molekulák uraciltartalmú DNS-hez kapcsolódnak, a létrejövő komplex nagyobb molekulatömeggel fog rendelkezni, és ezáltal az agaróz gélen feljebb fog megjelenni .
Eddigi eredmények
UNG-aktivitás-esszé és elektroforetikus mobilitáseltolódás-esszé
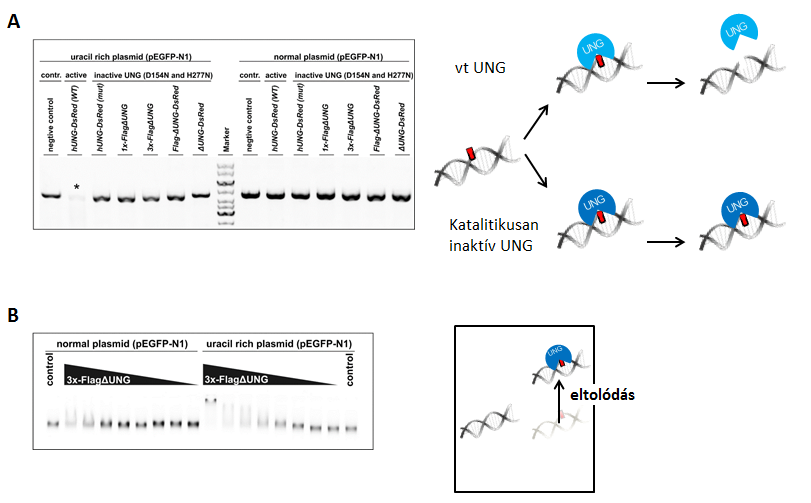
3. ábra A használt UNG-konstrukciók aktivitása (A) és uracilkötő-képessége (B)
A 3. ábra A paneljén az látható, hogy csak a hUNG2-DsRed WT (vad típusú UNG) -konstrukció aktív uracilgazdag plazmidon (csillaggal jelezve), amely nem rendelkezik a két pontmutációval. A 3x-FlagUNG jobban szeret az uracilgazdag DNS-hez kötni, mint a nem-uraciloshoz. Ez specifikus uracilkötési affinitást jelez (3. ábra B panel).
Kalibrációs egyenes dot blot-alapú mérésekhez
A katalitikusan inaktív 3xFlag-ΔUNG-ot használtam a dot blot-esszéhez. A jól definiált, magas uraciltartalmú log fázisú CJ236 [dut−, ung−] Escherichia coli törzsből izolált genomi DNS-t használtam standardként a mennyiségi meghatározások során: 4. ábra.
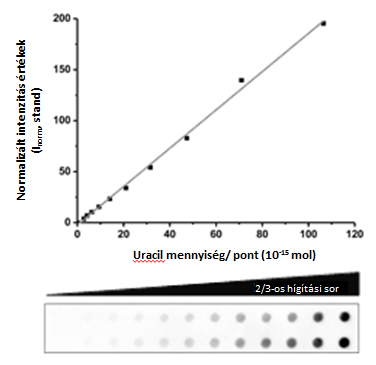
4. ábra Egy genomi uracil mennyiségi meghatározásra használt standard elrendezés
Különböző biológiai minták genomi uracil tartalmának meghatározása
Minden mérés esetében kalibrációs görbét vettünk fel az uracilszintek kiszámításához.
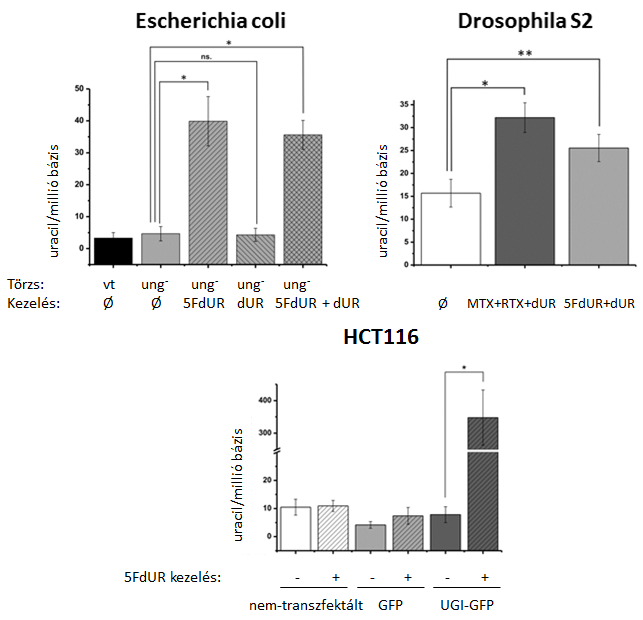
5. ábra Különböző fajok genomi uraciltartalmának meghatározása kemoterápiás szer jelenlétében, vagy anélkül (a csillag szignifikáns különbséget jelöl) [S4]
Az uracilszenzorunkat E. coli baktérium, S2 ecetmuslica sejtvonal és humán HCT116 sejtvonal DNS-beli uracilszintjének mennyiségi meghatározására használtuk. Ezen sejteket olyan timidilátszintézist gátló kemoterápiás szerekkel is kezeltük, mint az 5-fluorodezoxiuridin (5FdUR), a metotrexát (MTX), és a raltitrexed (RTX) és mértem a genom uracilszintjében bekövetkezett változást. A timidilát szintáz az az enzim, amely átalakítja a dezoxiuridin-monofoszfátot (dUMP) dezoxitimidin-monofoszfáttá (dTMP). Ez az egyedüli de novo dTMP szintézisútvonal az élőlényekben.
Várható impakt, további kutatás
A témában írt publikációim elismert folyóiratokban jelentek meg, különösen a Nucleic Acids Research-ben leközölt cikk, amely az itt leírt munka nagy részét lefedi, és amelynek impakt faktora 9,202 [S4]. Az új uracilkvantifikáló módszert bemutató közleményt a szakmai közvélemény érdeklődéssel fogadta: három külföldi kutató csoport is megkeresett minket (Norvégia, India, Egyesült Királyság). A közeljövőben különböző fejlődési állapotú (pete, lárva, …) teljes átalakulással fejlődő rovar genomi uraciltartalmát, és ennek fiziológiai következményét szeretném vizsgálni. Ezen rovarok nem kódolják az ung gént, sőt, néhány fejlődési stádiumban dUTPáz expresszió sincsen.
Saját publikációk, hivatkozások, linkgyűjtemény
Kapcsolódó saját publikációk listája (IF: impakt faktor, IC: független hivatkozások)
[S1] Szalkai B and Scheer I, Nagy K, Vértessy BG, Grolmusz V. The Metagenomic Telescope. PLoS One. 2014 Jul 23;9(7):e101605. (IF: 3,534, IC: 1)
[S2] Róna G, Marfori M, Borsos M, Scheer I, Takács E, Tóth J, Babos F, Magyar A, Erdei A, Bozóky Z, Buday L, Kobe B, Vértessy BG. Phosphorylation adjacent to the nuclear localization signal of human dUTPase abolishes nuclear import: structural and mechanistic insights. Acta Crystallogr D Biol Crystallogr. 2013 Dec;69(Pt 12):2495–505. (IF: 7,232, IC: 8)
[S3] Róna G, Pálinkás HL, Borsos M, Horváth A, Scheer I, Benedek A, Nagy GN, Zagyva I, Vértessy BG. NLS copy-number variation governs efficiency of nuclear import–case study on dUTPases. FEBS J. 2014 Dec;281(24):5463–78. (IF: 3,986, IC: 0)
[S4] Róna G, Scheer I, Nagy K, Pálinkás HL, Tihanyi G, Borsos M, Békési A, Vértessy BG. Detection of uracil within DNA using a sensitive labeling method for in vitro and cellular applications. Nucleic Acids Res. 2016 Feb 18;44(3):e28. ˙(IF: 9,202, IC: 0)
Linkgyűjtemény.
Hivatkozások listája.
[1] Lindahl T. Instability and decay of the primary structure of DNA. Nature. 1993 Apr 22;362(6422):709–15.
[2] Lozeron,H.A. and Szybalski,W. Incorporation of 5-fluorodeoxyuridine into the DNA of Bacillus subtilis phage PBS2 and its radiobiological consequences. J. Mol. Biol. 1967 Dec 14;30(2):277–90.
[3] Kiljunen S., Hakala K., Pinta E., Huttunen,S., Pluta P., Gador A.,Lonnberg H. and Skurnik M. Yersiniophage phiR1–37 is a tailed bacteriophage having a 270 kb DNA genome with thymidine replaced by deoxyuridine. Microbiology., 2005 151, 4093–4102.
[4] Yan N., O’Day E., Wheeler L.A., Engelman A. and Lieberman J. HIV DNA is heavily uracilated, which protects it from autointegration. Proc. Natl. Acad. Sci. U.S.A. 2011 108, 9244–9249.
[5] Pettersen H.S., Galashevskaya A., Doseth B., Sousa M.M., Sarno A., Visnes T., Aas P.A., Liabakk N.B., Slupphaug G., Saetrom P. et al. AID expression in B-cell lymphomas causes accumulation of genomic uracil and a distinct AID mutational signature. DNA Repair (Amst), 2015 25, 60–71.
[6] Muha V., Horvath A., Bekesi A., Pukancsik M., Hodoscsek B., Merenyi G., Rona G., Batki J., Kiss I., Jankovics F. et al. Uracil-containing DNA in Drosophila: stability, stage-specific accumulation, and developmental involvement. PLoS Genet. 2012 8,e1002738.
[7] Galashevskaya A, Sarno A, Vågbø CB, Aas PA, Hagen L, Slupphaug G, Krokan HE. A robust, sensitive assay for genomic uracil determination by LC/MS/MS reveals lower levels than previously reported. DNA Repair (Amst). 2013 Sep;12(9):699–706.
[8] Lari SU, Chen CY, Vertéssy BG, Morré J, Bennett SE. Quantitative determination of uracil residues in Escherichia coli DNA: Contribution of ung, dug, and dut genes to uracil avoidance. DNA Repair (Amst). 2006 Dec 9;5(12):1407–20.
[9] Horváth A, Vértessy BG. A one-step method for quantitative determination of uracil in DNA by real-time PCR. Nucleic Acids Res. 2010 Nov;38(21):e196.
