|
|
BMe Kutatói pályázat |
|
Oláh György Doktori Iskola
Alkalmazott Biotechnológia és Élelmiszertudományi Tanszék
Témavezető: Dr. Vértessy Beáta, Dr. Tasnádi Gábor
Aminok biokatalitikus aszimmetrikus szintézise reduktív aminálással
A kutatási téma néhány soros bemutatása
A doktori kutatás általános célja egy modern, természetes katalizátorokon (enzimeken) alapuló szintézismódszer fejlesztése és alkalmazása gyógyszerkutatási szempontból fontos vegyületek, elsősorban királis aminok előállítására. A kutatás ipari relevanciáját a nagyszámú gyógyszermolekulában fellelhető királis amin szerkezeti elem adja, melynek előállítása jelentős szintetikus kihívást jelent. A doktori kutatás eredményeként egy olyan enzimkészlet és kapcsolódó tudásbázis jön létre, mely királis aminok környezetkímélő és hatékony előállítását teszi lehetővé.
A kutatóhely rövid bemutatása
A kutatás a BME Alkalmazott Biotechnológia és Élelmiszertudományi Tanszékén működő Biostruct Labor és a Servier Kutatóintézet együttműködésében valósul meg. A szerkezeti biológiában kiemelkedő szaktudással rendelkező Biostruct Laborban kivitelezhető az új enzimszekvenciák bioinformatikai azonosítása és elemzése, a fehérjék fermentációval történő előállítása és tisztítása, valamint a szerkezetek modellezése és meghatározása. A Servier Kutatóintézet ipari partnerként nemzetközi szinten is kiemelkedő infrastruktúrát kínál a kész enzimpanelek, illetve legyártott enzimek tesztelésére, a megfelelő szubsztrátok szintézisére, az analitikai módszerek kidolgozására, illetve a szintetikus alkalmazások vizsgálatára.
A kutatás történetének, tágabb kontextusának bemutatása
A modern gyógyszerjelölt molekulák szerkezete egyre összetettebb, gyakran több kiralitáscentrumot is tartalmaz, melyek gazdaságos szintézise hatalmas kihívás a szintetikus vegyészek számára. Ezért az eljárásfejlesztés során egyre nagyobb jelentőségük van az olyan technológiáknak, melyek a szintetikus lépések számát csökkentik, azok szelektivitását és összességében hatékonyságát növelik. A biokatalízis ezen célok megvalósulásához nagymértékben hozzá tud járulni, az enzimek fenntartható előállításának és felhasználásának, valamint kivételes szelektivitásának köszönhetően (1. ábra). A biokatalízis az utóbbi két évtizedben rendkívüli fejlődésen ment át; laboratóriumi módszerből iparilag releváns technológiává lépett elő. Ez a fejlődés elsősorban az enzimek tervezhetőségének és optimalizálhatóságának köszönhető, melyeket a biológiai tudományok robbanásszerű fejlődése és az irányított evolúció technológiája hajtanak. Míg korábban az enzimatikus ipari folyamatokat jelentősen limitálta, hogy az eljárás paramétereit az enzim tulajdonságaihoz kellett igazítani, manapság sok esetben az enzimet alakítják az eljárás körülményeihez és a szubsztrát(ok)hoz (enzimmérnökség). Ennek eredményeképpen egyre több ipari enzimes eljárás születik, sok közülük gyártási méretben[1].

1. ábra: A biokatalízis előnyei ipari gyártási folyamatokban
A becslések szerint a gyógyszerkészítmények mintegy 40%-a tartalmaz királis amin építőelemet, ezért is nagy az érdeklődés enantiomertiszta formában történő előállításuk iránt. Számos enzimatikus megoldás ismert királis aminok szintézisére, melyek közé a közelmúltban több újabb enzimcsalád is bekerült. Az imin-reduktázok (IRED) egy alcsoportjáról, az ún. reduktív aminázokról (RedAm) kimutatták, hogy az imin képződését is katalizálják, aminek köszönhetően közel sztöchiometrikus mennyiségű aminpartner használata is elegendőnek bizonyul magas konverziók eléréséhez[2]. Ez a fajta reaktivitás két molekula (karbonil és amin) kapcsolását teszi lehetővé királis szekunder amin vegyületeket eredményezve. Megfelelő szubsztrátokkal reduktív amináz aktivitást mutatnak más kevésbé ismert enzimcsaládok tagjai is, mint például a ketimin reduktázok (KIRED) vagy az opin dehidrogenázok (ODH)[3].
A kutatás célja, a megválaszolandó kérdések
A kutatás célja egy reduktív amináz enzimgyűjtemény létrehozása és funkcionális jellemzése. Az új enzimes paneleket részben ismert enzimekből, részben bioinformatikai módszerekkel azonosított új enzimekből állítjuk össze.
Az elsődleges célreakció különböző aminosav-származékok amincsoporton keresztüli kapcsolása változatos karbonilvegyületekkel, elsősorban ketosavakkal (2. ábra). Az így keletkező királis szekunder amin dikarbonsav-származékok a természetben is előforduló opin típusú vegyületek[4] analógjai, melyek az oldalláncban elhelyezkedő funkciós csoportokon keresztül vagy a karbonsavfunkció módosításával további származékokká alakíthatóak. Ezek értékes építőelemei lehetnek bioaktív molekuláknak, mint például a peptidomimetikumoknak. Az ilyen vegyületekre példák a 3. ábrán látható ACE2 gátlószerek.
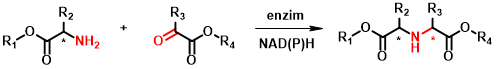
2. ábra: A kutatás fókuszában álló reduktív aminálási reakció általános egyenlete
Az egyedi
gyógyszermolekula építőelemekhez való igazítás megkívánja, hogy ismerjük az
enzim aktív centrumát és kötőzsebeit. Ezen ismeretek felhasználásával
módosításokat tudunk végrehajtani az enzim szerkezetében, hogy változatos,
további kémiai kapcsolásokat megengedő királis aminvegyületeket tudjunk
szintetizálni magas sztereoszelektivitással. A kutatás célja fókuszált
variánskönyvtárak számítógépes tervezése, valamint a tervezett variánsok
gyors és egyszerű előállítására és tesztelésére alkalmas módszer kifejlesztése
is. Ennek segítségével a létrehozott enzimpanelek gyorsan tovább bővíthetők,
illetve testre szabhatók lehetnek egy adott szintetikus probléma megoldására.
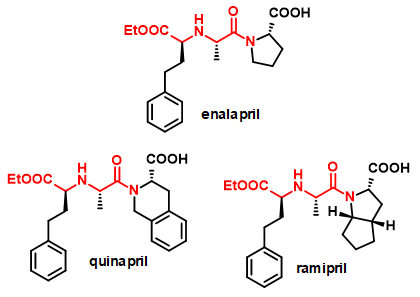
3. ábra:
Királis szekunder amin dikarbonsav építőelemet tartalmazó gyógyszermolekulák
Az enzimgyűjteményből kiválasztott, a modell szubsztrátokat leghatékonyabban átalakító enzimekkel a reakciókörülményeket is kívánjuk optimalizálni preparatív méretű átalakítások megvalósítása érdekében. Ez a méretnövelési lépés kiemelten fontos ezen új enzimes eljárások ipari alkalmazása szempontjából.
Módszerek
A kutatás a reduktív amináz enzimgyűjtemény felépítését átfogó módon, többféle megközelítést alkalmazva valósítja meg. A legkézenfekvőbb módszer az irodalomban már korábban leírt enzimek tesztelése a modell szubsztrátokkal. Az enzimek szubsztráttoleranciáját kihasználva bővíthető azon molekulák köre, amelyek hatékony átalakítására egy adott enzim használható. Ezzel a módszerrel a kutatás során tesztelünk kereskedelmi forgalomban kapható enzimeket, például az angol Prozomix cég imin-reduktáz (IRED) gyűjteményét[5], illetve más, kevésbé kutatott enzimcsaládok ismert tagjait, például ketimin-reduktázokat (KIRED) és opin-dehidrogenázokat (ODH). Az enzimek szubsztrátköre általában csak limitáltan bővíthető, ezért egy szintetikus probléma megoldásához további módszerekre is szükség van. Ha egy enzim nem mutat aktivitást egy szubsztráton, két megoldás áll a rendelkezésre (4. ábra).
Az első a természetben rejlő sokféleség (biodiverzitás) kihasználása. Szekvenciahasonlóság alapján bioinformatikai módszerekkel genomi és metagenomikai adatbázisokban új enzimek találhatók, amelyek hasonló aktivitással, de más szubsztrátkörrel rendelkeznek. Ez utóbbi adatbázisok különösen is értékes tulajdonságokkal rendelkező enzimek forrásai is lehetnek, hiszen gyakran extrém környezetből származó minták genetikai információit tartalmazzák[6]. Az ezekben kódolt fehérjék toleránsabbak az ipari felhasználáskor előforduló nem természetes körülményekkel szemben (magas hőmérséklet, szerves oldószerek stb.). Egy akadémiai együttműködés keretében a kutatás során környezeti metagenomokból új szekvenciákat azonosítunk, majd a kódolt enzimeket előállítjuk és karakterizáljuk.
A második lehetőség az adott enzim aktivitásának javítása az aminosav szekvenciájának módosításával (enzimmérnökség). Ezen a megközelítésen alapul a Nobel-díjas irányított evolúció módszere, amely során iteratív módon végzett nagy mennyiségű mutáció eredményeként egy jelentősen megnövelt aktivitású enzimet kaphatunk[7]. Ez a technológia azonban nagyon idő- és erőforrásigényes. Amennyiben rendelkezésre áll információ az enzim szerkezetéről, illetve működésének mechanizmusáról, racionális módon is tervezhetők aktivitást javító mutációk, így jelentősen csökkenthető a tesztelendő variánsok száma[8]. A kutatás során kiválasztott enzimeken számítógépes módszerekkel tervezett mutációkat hajtunk végre aktivitásuk növelése, illetve szubsztrátpreferenciájuk megváltoztatása érdekében.
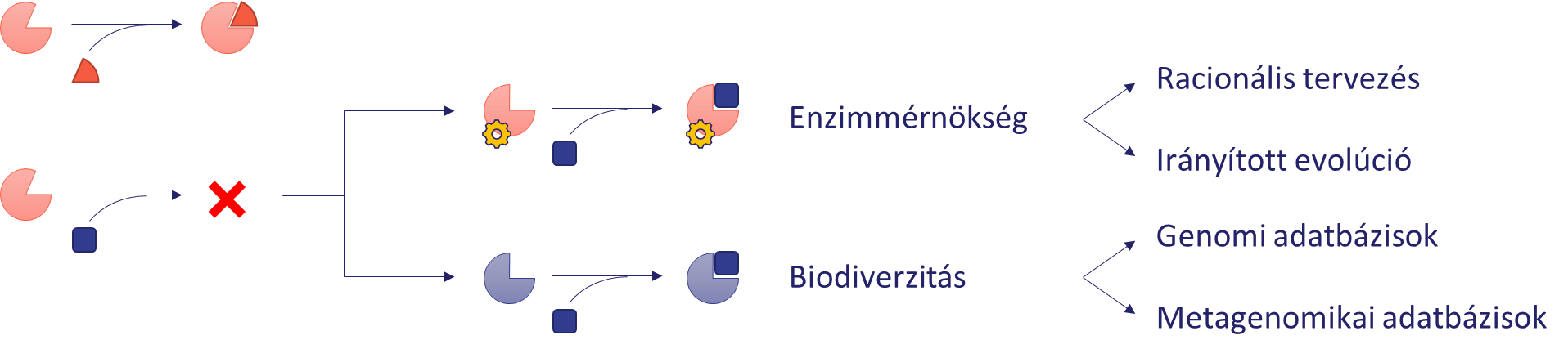
4. ábra: Stratégiák egy új szubsztrát átalakítására képes enzim előállítására
Egy mutáció azonban ritkán eredményez elégséges aktivitásnövekedést, így racionális tervezés esetén is szükséges kisebb variánskönyvtárak tesztelése. A kutatás során egy olyan módszert is fejlesztünk, amely alkalmas közepes méretű (10–100-as nagyságrendű) variánskönyvtárak gyors előállítására és tesztelésére. Ebben a folyamatban limitáló tényező a módosított enzimek bakteriális expressziója és megtisztítása. Ezen lépés felgyorsítása érdekében a kutatás során sejtmentes fehérjeexpressziós rendszert alkalmazunk. E módszer segítségével a kezdeti aktivitástesztekhez szükséges kis mennyiségű enzim gyorsan és egyszerűen előállítható, nagyban meggyorsítva a folyamatot[9]. Ahhoz, hogy a sejtmentes expressziós rendszer enzimvariánsok vizsgálatára is alkalmas legyen, egy innovatív, új módszert fejlesztünk a variáns DNS-szekvenciák sejtmentes (pusztán PCR-alapú) előállítására. Ezt a folyamatot a sejtmentes expresszióval összekapcsolva az ipari hasznosításhoz szükséges enzimmérnökség is gyorsan és akár egy kémiai laboratóriumban is megvalósítható.
Eddigi eredmények
A kereskedelmi forgalomban kapható IRED-ek vizsgálatának kiindulási alapja az irodalomban leírt α-ketoésztereken mutatott aktivitásuk, amely N-szubsztituált aminosavszármazékok szintézisét teszi lehetővé[10]. Kutatásunk során a leírt szubsztrátok körét bővítettük α-ketosav, illetve aminoészter-típusú molekulákkal (5. ábra). A teljes, 384 enzimet számláló kollekcióból többféle szubsztrátkombináció tesztelése után 18 enzimet választottunk ki, amelyeket tisztított formában is vizsgáltunk, és optimalizáltuk a reakciókörülményeket.
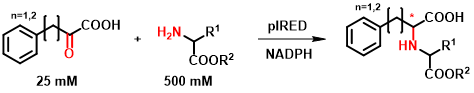
5. ábra: Az új, IRED-ek által katalizált reakció általános egyenlete
A KIRED-ek és az ODH-k α-ketosavak reduktív aminálását képesek elvégezni, az előbbiek primer aminokkal[11], az utóbbiak α-aminosavakkal[12]. 3 irodalomban leírt KIRED enzim vizsgálata során azt találtuk, hogy az enzimcsalád kiemelkedő aktivitást mutat α-ketosav/α-aminoészter szubsztrátkombinációval (6. ábra A). A legjobban teljesítő enzimmel (a Rattus norvegicus-ból származó RnKIRED) folytattunk részletesebb vizsgálatokat, és részletesen feltérképeztük szubsztrátkörét (6. ábra B). E folyamat részeként optimalizáltuk a reakciókörülményeket (pH, hőmérséklet, szubsztrátkoncentráció, enzimkoncentráció), és folyamatban van a keletkező termékek abszolút konfigurációjának meghatározása. Érdekes megfigyelés volt, hogy a szubsztrátok mérete befolyásolja az újonnan létrejövő sztereocentrum konfigurációját: nagyméretű ketosav és kisméretű aminoészter reakciójában S-szeletivitást, míg kisméretű ketosav és nagyméretű aminoészter esetében R-szelektivitást mutat az RnKIRED (6. ábra C).
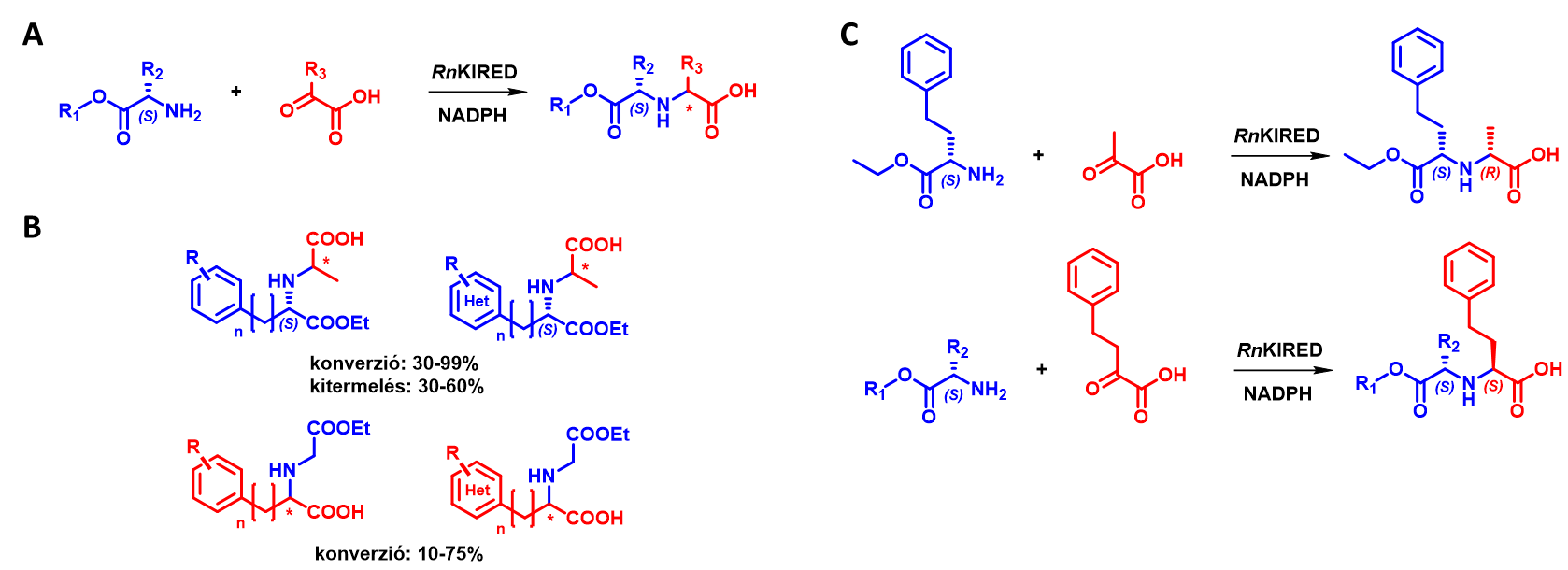
6. ábra: A
Az RnKIRED által katalizált új reakció általános egyenlete. B Az
RnKIRED szubsztrátköre
C Az RnKIRED sztereoszelektivitásának változása különböző térkitöltésű
szubsztrátokkal
Az általunk előállított és vizsgált 5 ismert ODH-enzim csak a leírt ketosav/aminosav kombinációban mutattak aktivitást, ketoészterrel vagy aminoészterrel nem. E kevéssé kutatott enzimcsaládot azonban tovább is vizsgáltuk, hiszen az általuk katalizált reakció nagyon közel áll az általunk definiált célreakcióhoz. Hőforrásból származó metagenomikai adatbázisból 10 új ODH-szekvenciát azonosítottunk és 6 enzim karakterizálását valósítottuk meg. Ezen teljesen új metagenomikus opin-dehidrogenázok (mODH-k) magasabb hőtűrésűnek bizonyultak, mint az irodalomban eddig ismert ODH-k. Az mODH-k az irodalomban leírtakhoz képest eltérő szubsztrátokon mutattak aktivitást, jellemzően negatív töltésű poláros aminosavakat tudtak átalakítani. Tipikus reakciópartner volt az L-aszparaginsav (7. ábra B), de nem kanonikus aminosavakat is elfogadtak, mint pl. L-ciszteinsav. Egy kiválasztott metagenomikus enzimmel több preparatív méretű (~100 mg) transzformációt is elvégeztünk. Ennek eredményeképp három nagymértékben funkcionalizált királis szekunder aminszármazékot sikerült előállítani és kémiailag karakterizálni (7. ábra C).
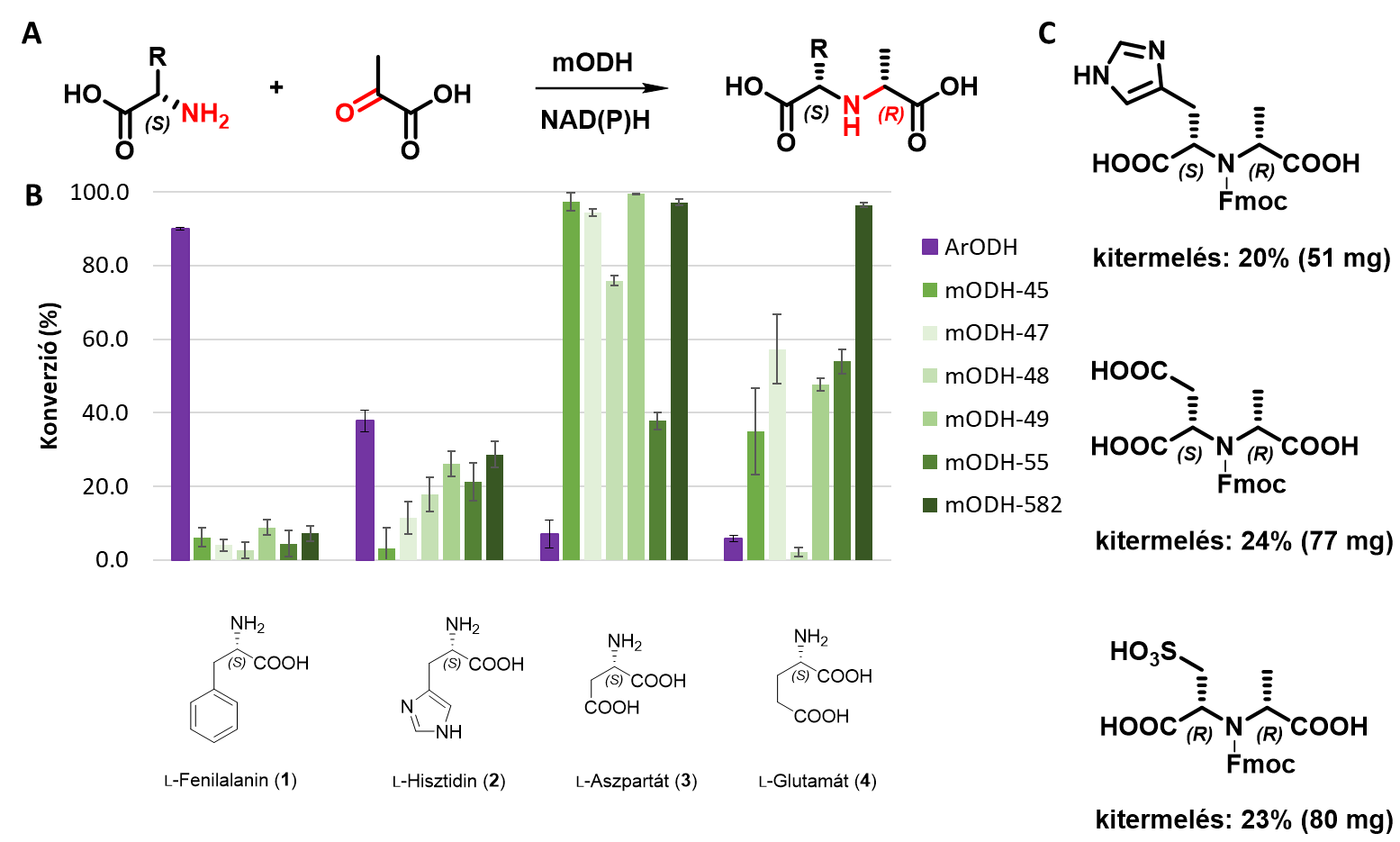
7. ábra: A Az mODH-k által katalizált reakció általános egyenlete. B Az mODH-k szubsztrát preferenciája, összehasonlítva a referencia ArODH-val. C mODH-k felhasználásával szintetizált új molekulák.
Az mODH-k eddig példátlan aminosavpreferenciáját szerkezeti modellek segítségével értelmeztük. Az aktív zseb részletesebb vizsgálata rávilágított arra, hogy egyes aminosav-oldalláncok kémiai karaktere, valamint térbeli közelsége is meghatározó lehet. Ebből kiindulva az aktív zsebben végrehajtottuk két pozíció egyidejű módosítását, amivel sikerült megfordítani az ODH-enzimek szubsztrátpreferenciáját: a metagenomikus enzim dupla mutáns változata apoláris aminosavakkal, míg a referencia enzim dupla mutáns verziója negatív töltésű poláris aminosavakkal működött (8. ábra).
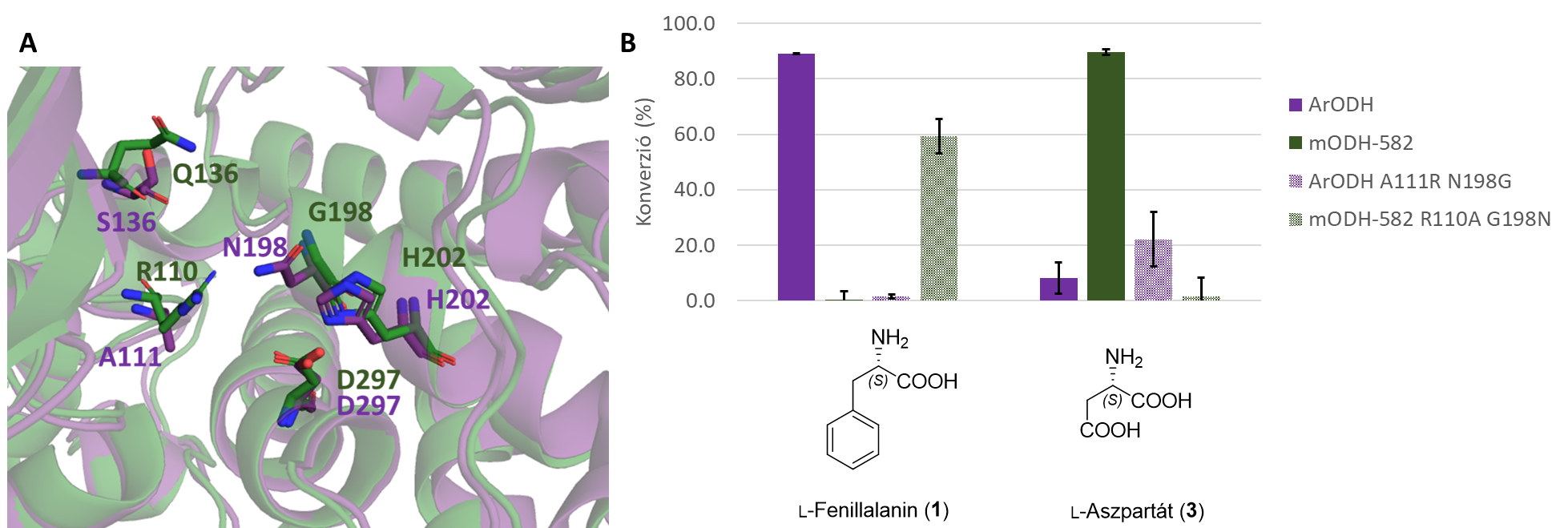
8. ábra: A Egy mODH (zöld) és a referencia ArODH (lila) aktívzsebeinek összehasonlítása a legfontosabb aminosav-oldalláncok jelölésével. B ODH-k szubsztrátpreferenciájának felcserélődése két racionálisan tervezett pontmutáció hatására.
A kutatás során sikeresen felállítottunk egy sejtmentes fehérjeexpressziós rendszert, amely PCR-reakcióban előállítható lineáris templát-DNS felhasználásával kis mennyiségű enzim termelésére és tisztítására alkalmas. Ezzel új enzimek, illetve enzimvariánsok gyors tesztelése valósítható meg. A variáns DNS-szekvenciák sejtmentes előállítására innovatív módszert dolgoztunk ki, amit ODH- és KIRED-mutánsokon validáltunk. Utóbbi esetben egy számítógépes módszerrel tervezett 60 tagú variánskönyvtár előállítása is megvalósult.
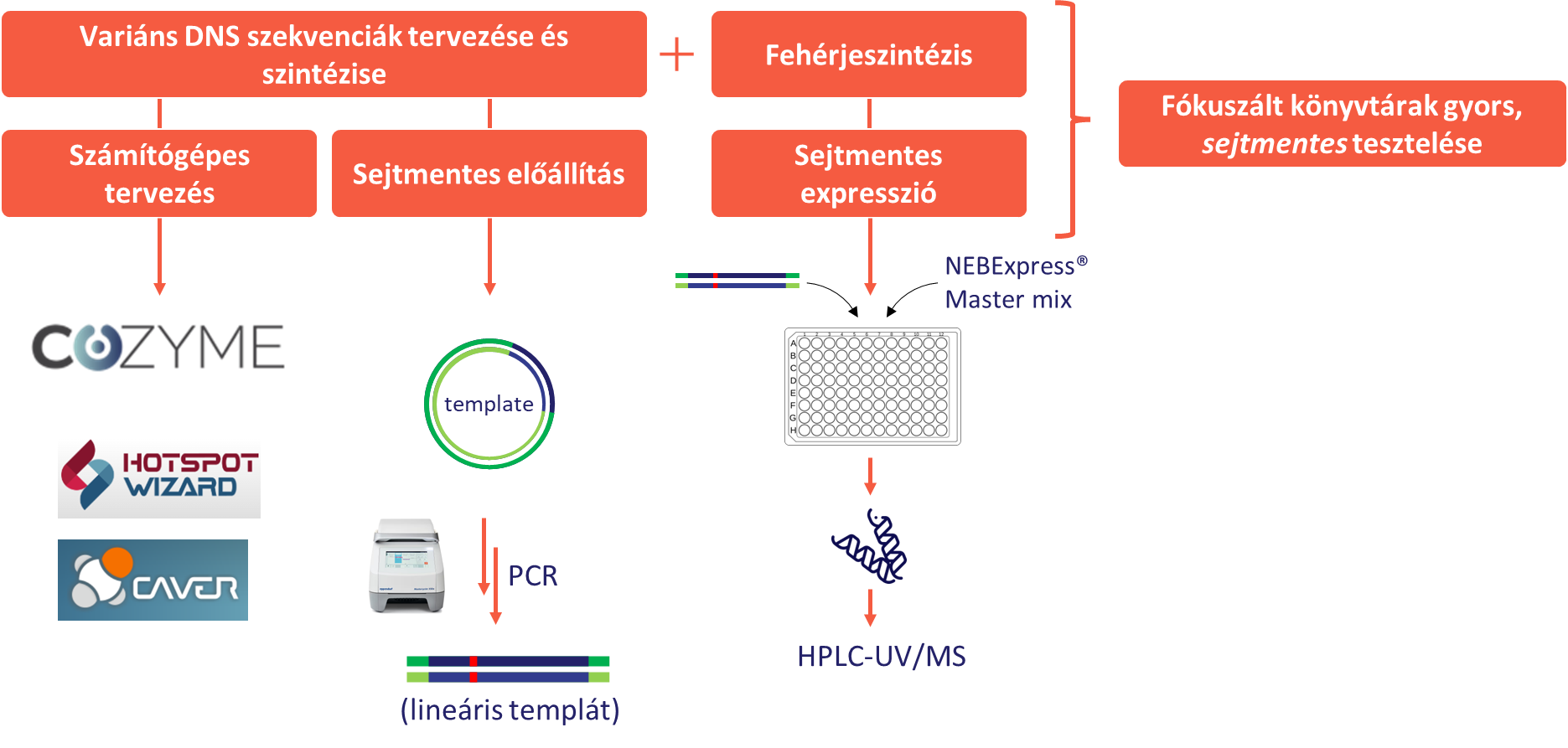
9. ábra: A sejtmentes fehérjemérnökség koncepciója
Várható impakt, további kutatás
Az eddigi kutatás jelentős eredményeket hozott a reduktív aminálásra alkalmas enzimes eljárások fejlesztésében mindhárom különböző megközelítés esetében. Eredményeink jelentős hatással vannak a Servier-nél folyó biokatalitikus aktivitások bővítésére és infrastrukturális fejlesztésére. Az IRED és KIRED enzimcsaládok esetében kibővítettük ezek hasznosíthatósági körét új szubsztrátok bevonásával. Az ODH enzimcsaládot környezeti metagenomból származó új tagokkal bővítettük, ezeket az enzimeket részletesen karakterizáltuk, és demonstráltuk szintetikus hasznosíthatóságukat. Az általunk kifejlesztett sejtmentes mutagenezis és expressziós módszer lehetővé teszi a már rendelkezésre álló enzimek házon belüli továbbfejlesztését is (9. ábra). További célunk ennek a módszernek a kiterjesztett alkalmazása új enzimcsaládok vizsgálatára (amin dehidrogenázok), valamint az RnKIRED enzim fejlesztésére. A fejlesztés célja egy ACE2-gátló gyógyszerekben előforduló kulcsintermedier sztereoszelektív előállítása.
Saját publikációk, hivatkozások
Kapcsolódó saját publikációk listája:
András Telek, Zsófia Molnár, Beáta G Vértessy, Gábor Tasnádi: Opine dehydrogenases, an underexplored enzyme family for the enzymatic synthesis of chiral amines. Biotechnol. Bioeng., 2023, 120, 2793–2808.
András Telek, Zsófia Molnár, Kristóf Takács, Bálint Varga, Vince Grolmusz, Gábor Tasnádi, Beáta G Vértessy: Discovery and biocatalytic characterization of opine dehydrogenases by metagenome mining. Appl. Microbiol. Biotechnol. 2024, 108, 1–16.
Hivatkozások listája:
[1] E. L. Bell et al. Nat. Rev. Methods Primers 2021, 1, 46−67.
[2] M. D. Patil et al. ACS Catal. 2018, 8, 10985−11015.
[3] J. H. Schrittwieser et al. Adv. Synth. Catal. 2015, 357, 1655–1685.
[4] J.F. Hyslop et al. J Biotechnol. 2019, 293, 56–65.
[5] J. R. Marshall et al. Nat. Chem. 2021, 13, 140−148.
[6] S. L. Robinson, J. Piel, S. Sunagawa Nat. Prod. Rep. 2021, 38, 1994–2023.
[7] J. L. Porter, R. A. Rusli, D. L. Ollis ChemBioChem 2016, 17, 197–203.
[8] A. Phintha, P. Chaiyen Chem. Catal. 2022, 2, 2614–2643.
[9] J. Rolf, K. Rosenthal, S. Lütz Catalysts 2019, 9, 190–207.
[10] P. Yao et al. Angew. Chemie Int. Ed. 2021, 60, 8717–8721.
[11] J. F. Hyslop et al. Angew. Chemie 2018, 130, 14017–14020.
[12] Y. Kato, H. Yamada, Y. Asano J. Mol. Catal. B Enzym. 1996, 1, 151–160.
A C1580174 számú projekt a Kulturális és Innovációs Minisztérium Nemzeti Kutatási Fejlesztési és Innovációs Alapból nyújtott támogatásával, a NVKDP-2021 pályázati program finanszírozásában valósult meg.
